作者简介: 梁红玲(1984-),女,湖南耒阳人,主治医师,在读博士研究生,主要从事乳腺肿瘤多学科综合治疗的临床和基础研究工作。
目的 比较分析Sanger测序法和Snapshot 法检测乳腺癌BIM缺失多态性的一致性。方法 共收集336例乳腺癌患者手术标本制备成石蜡包埋的蜡块,提取DNA后采用Sanger测序法和Snapshot 法检测乳腺癌患者中BIM基因缺失多态性突变率。采用卡方检验分析BIM缺失多态性在不同临床特征患者中的比例情况。采用配对卡方检验和κ系数检验分析两种方法的一致性。结果 Sanger测序法检测出39例患者BIM基因缺失,阳性率为11.61%;Snapshot法检测出47例患者BIM基因缺失,阳性率为13.99%。Sanger测序法检测出的BIM突变状态在不同年龄阶段( χ2=6.242, P=0.044)和孕激素表达水平( χ2=4.070, P=0.044)的乳腺癌中差异有统计学意义。配对卡方检验显示两种检测方法差异无统计学意义( P=0.152),Kappa检验结果表明两种方法的一致性较好(吻合系数κ=0.695, P=0.000)。结论 在中国乳腺癌患者中BIM缺失多态性的比例约为11.61%。Sanger测序法和Snapshot 法检测乳腺癌BIM缺失多态性具有较好的一致性,可以相互补充。
Objective The role of this essay is to explore the cocordance of Sanger sequencing and Snapshot methods for detecting the deletion polymorphism of BIM gene in breast cancer.Methods Surgical cancer tissue samples of a total of 336 cases with breast cancer were collected and preserved as formalin fixed paraffin embedded wax blocks. Genomic DNA was extracted and BIM gene deletion polymorphic in these breast cancer patients were detected by Sanger sequencing and Snapshot methods. Chi-square test with cross-tabular data was used to analyze the proportion of BIM deletion polymorphisms in patients with different clinical characteristics. Chi square test and Kappa coefficient test were used to detect the concordance degree of the two methods.Results In these 336 cases of breast cancers, 11.61% (39/336) cases were tested as positive with BIM deletion by Sanger sequencing. 13.99% (47/336) patients were positive with BIM deletion porlymorphism by Snapshot method. The Sanger sequencing method detected statistically significant differences in the expression of BIM mutations in breast cancer at different ages ( χ2=6.242, P=0.044)and progestrone receptor levels( χ2=4.070, P=0.044). The McNemar test showed the two methods had no statistical difference in the positivity of BIM gene deletion polymorphism ( P=0.152). Kappa coefficient test also indicated concordance between the two methods (κ=0.695, P=0.000).Conclusions In this Chinese cohort of breast cancers, the BIM gene deletion polymorphism is around 11.61%. Sanger sequencing and Snapshot methods had consistency and might be complementary in the detection of BIM deletion polymorphism in breast cancer.
BIM (Bcl-2 interacting mediator of cell death) 全名为BCL-like 11, 是凋亡分子Bcl-2家族成员之一, 在乳腺癌、肺癌、骨肉瘤和黑色素瘤中起着促进细胞凋亡的重要作用[1, 2]。近期发现BIM缺失多态性与EGFR突变阳性的非小细胞肺癌等肿瘤靶向抑制剂的疗效不佳相关[3]。更多的研究发现BIM缺失多态性是非小细胞肺癌、慢性粒细胞白血病、急性粒细胞白血病以及乳腺癌不良预后的肿瘤分子标志物[4, 5, 6, 7, 8]。
目前基因测序、qPCR基因分型技术、酶切电泳方法、基因芯片等是基因多态性检测的主要方法, 而基因测序是国际公认的最标准、最准确的方法[9, 10]。Snapshot方法是基因分型技术中的一种, 其特点为检测通量高; 基因芯片也有高通量的优势, 但由于目前检测及评价标准不统一, 加上各实验室硬件、软件以及操作人员的不同, 往往造成结果重复率不高。为提高准确性和检出率, 疾病易感基因检测可能需根据情况采用多种方法相结合的手段, 如测序法和其他基因分型技术共同完成[11]。本研究利用Sanger测序法和Snapshot法检测乳腺癌患者BIM基因缺失多态性, 以了解BIM基因缺失多态性在乳腺癌中的发生率, 并比较两种检测方法在诊断乳腺癌患者BIM基因缺失多态性突变的可行性和吻合度情况。
本研究回顾性分析2008年1月到2011年8月期间在广州医科大学附属肿瘤医院行乳腺癌改良根治或保乳手术336例患者的原发灶癌组织石蜡标本和完整临床资料。纳入标准:(1)病理组织学明确为浸润性导管癌或以浸润性导管癌为主, 伴其他特殊组织学类型; (2)采用乳腺癌改良根治或者保乳手术完整切除原发肿瘤; (3)患者年龄≥ 15岁; (4)依据NCCN指南接受规范化的乳腺癌综合治疗; (5)研究中所涉及的石蜡标本及临床资料均经由患者知情同意, 并经由医院伦理委员会批准。石蜡包埋的手术标本采用Qiagen DNA提取试剂盒提取DNA。简述步骤为:二甲苯脱蜡, 无水乙醇脱二甲苯, 蛋白水解酶混匀, 56℃水浴锅孵育过夜; 24小时后, 金属浴1小时, 冷却后迅速加入无水乙醇, 13 000 rpm反复离心5次; 70℃金属浴放置20~30 min, 酒精挥发完全后13 000 rpm离心1.5 min; 将DNA保存于-20℃备用。
测序试剂采用BigDye Terminator3.1 及qPCR Mix反应液, 均购自美国Life Tech 公司; 探针和引物在广州英伟创津公司合成。DNA和RNA抽提试剂盒均购自德国Qiagen公司。引物(BIM_DEL_F: 6-FAMGCTAACTCAACAAACCCATCAGAAC; IM_ DEL_R:AGCCAGTAAATATAAATCCAAAGCA)利用Primer express3.0软件自行设计, 委托华大基因公司合成; 主要仪器为美国ABI 3730XL测序仪和ABI 7500Fast 荧光定量PCR仪器。结果采用Chromas2.31软件和Gene Scan Genotyper3.5软件进行分析。
(1)按照实验需要的反应体系和用量计算各组分的加量配置Mix液。
(2)用Millipore水稀释合成好的引物(BIM_DEL_F:6-FAMGCTAACTCAACAAACCCATC AGAAC; BIM_DEL_R:AGCCAGTAAATATAAATCC AAAGCA)。
(3)配制PCR反应体系:每个25 μ L的PCR反应包括1 μ L (20 ng/μ L)模板DNA, 12.5 μ L Premix Ex Taq HS酶(大连TAKARA公司), 1 μ L正向和反向引物混合物, 10.5 μ L ddH20。
(4)PCR反应条件:初始变性94℃ 5 min, 变性94℃ 30 sec, 退火55℃ 30 sec, 72℃延伸1 min, 35个循环, 72℃延伸7 min。将扩增的PCR产物经2%琼脂糖凝胶电泳检测目的片段符合条件后, 纯化产物。
(5) PCR产物采用BigDyeTeminator V3.1 按操作说明书进行标记及纯化, 然后在ABI-3730测序仪上进行双向测序, 测序结果用Chromas2.31软件分析。
(1)引物设计和合成:BIM_DEL_F: 6-FAMGC TAACTCAACAAACCCATCAGAAC; BIM_DEL_R:
AGCCAGTAAATATAAATCCAAAGCA。
(2)DNA提取:按试剂说明书提取基因组DNA, 测OD值, 并配成10 ng/μ L。
(3)PCR扩增:配置总反应体系10 μ L, 含Premix Ex Taq Hot Start Version 5 μ L(大连TAKARA公司), DNA模板0.5 μ L, 2 μ M上下游引物各0.5 μ L, ddH2O 3.5 μ L。扩增条件为:94℃预变性3 min, 94℃变性30 sec, 55℃退火30 sec, 72℃延伸1 min, 共30个循环。
(4) 基本扫描分析取荧光标记的PCR产物1 μ L, 加69 μ L水以1∶ 70稀释, 取1 μ L与0.5 μ L Genescan-500 LIZ Size Standard及9 μ L去离子甲酰胺混合, 在ABI-3730 DNA序列分析仪中进行毛细管电泳, Gene Scan Genotyper3.5软件自动分析收获的数据。
应用统计学软件SPSS 20.0进行统计分析。采用配对卡方检验和Kappa检验分析两种方法检测结果的差异性和吻合度。P≤ 0.05定义为两种检测方法差异有统计学意义(κ ≥ 0.7, 表示吻合度较强; 0.7 > κ ≥ 0.4表示吻合度一般; κ ≤ 0.4表示吻合度较弱)。采用卡方检验分别分析两种检测方法测得BIM缺失多态性在336例乳腺癌患者不同临床特征患者中的比例情况。
Sanger测序法和Snapshot法检测336例乳腺癌标本BIM缺失多态性的样图见图1和图2。
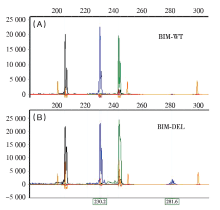 | 图2 Snapshot 检测法 PCR产物片段的毛细管电泳基因扫描峰图 (A) 野生型病例, (B)缺失型病例, 黑色/蓝色/绿色波峰分别是EGFR-EX19/HER2-EX20/EGFR-EX19片段 |
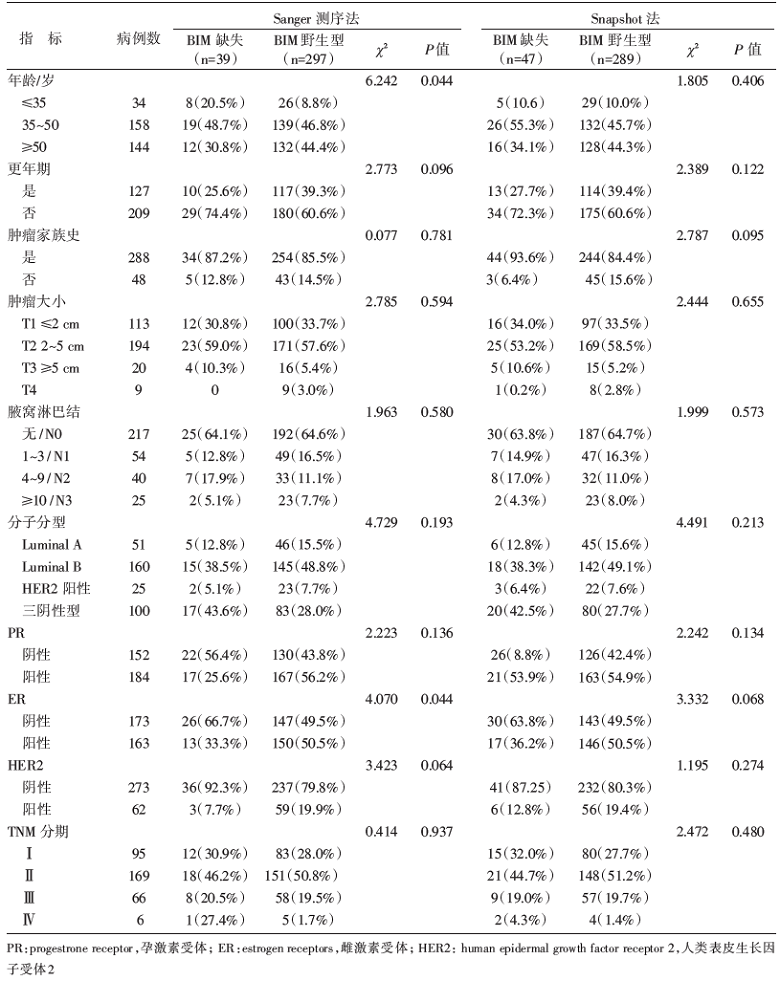
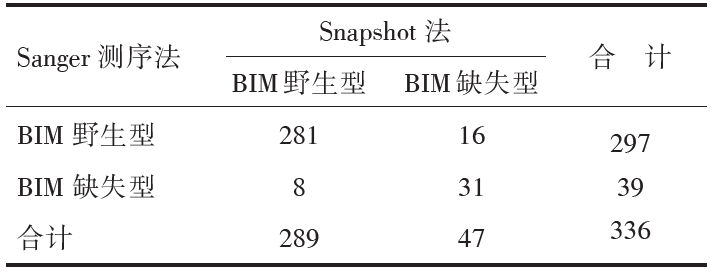
Sanger直接测序法发现39例患者为BIM基因缺失, 阳性率为11.61%; Snapshot 法检测法发现47例患者BIM基因缺失, 阳性率为13.99%。两种方法共同测出BIM基因缺失的为31例, 两种方法均测出为野生型的为281例; 另外, Snapshot 法检测出BIM缺失突变而Sanger测序法检测为野生型的为16例, Snapshot 法检测为BIM基因野生型而Sanger测序法检测为缺失突变的为8例(见表2)。其中, Sanger直接测序法测得BIM基因缺失突变在乳腺癌不同分子分型的例数和百分比分别是Luminal A型5例(12.8%), Luminal B型15例(38.5%), HER2阳性型2例(5.1%), 三阴性型17例(43.6%); Snapshot 法测得BIM基因缺失突变在不同分子分型的例数和百分比分别是Luminal A型6例(12.8%), Luminal B型18例(38.3%), HER2阳性型3例(6.4%), 三阴性型20例(42.5%)。Sanger直接测序法测得BIM基因缺失突变在不同分期中的例数和百分比分别是Ⅰ 期12例(30.9%), Ⅱ 期18例(46.2%), Ⅲ 期8例(20.5%), Ⅳ 期1例(27.4%); Snapshot 法测得BIM基因缺失突变在不同分期中的例数和百分比分别是Ⅰ 期15例(32.0%), Ⅱ 期21例(44.7%), Ⅲ 期9例(19.0%), Ⅳ 期2例(4.3%)(见表1)。两种方法测得BIM基因缺失突变在不同ER、PR、HER2表达情况, 不同肿瘤大小及淋巴结转移个数中的例数和百分比详细见表1。其中Sanger测序法检测到BIM突变状态在不同年龄阶段(χ 2=6.242, P=0.044)和不同PR表达水平(χ 2=4.070, P=0.044)的乳腺癌中差异有统计学意义。
| 表2 Sanger测序法和Snapshot法检测乳腺癌BIM基因缺失突变病例数情况 |
配对卡方检验结果显示两种检测方法检测BIM的缺失突变率的差异无统计学意义(P=0.152); Kappa检验结果提示两种方法的结果一致(吻合系数κ =0.695, P=0.000)。认为两种检测方法检测乳腺癌BIM缺失多态性具有较好的一致性。
BIM为BCL-2蛋白家族成员, 是活性最强的促凋亡蛋白之一。BCL-2蛋白家族分为促凋亡和抗凋亡两类功能分子。BIM含有BH3结构域, 该结构域含有重要的促凋亡功能。BIM与BCL-2蛋白家族其他抗凋亡成员如BCL2、BCL2L1/BCL-X(L)和MCL1相互作用, 相互调节, 介导细胞凋亡。BIM基因经转录后形成多种转录本, 进而翻译形成多种蛋白异构体, 通常一个蛋白异构体中只有4号外显子或3号外显子编码信息, 两者不会共存。4号外显子编码含BH3结构域具有促凋亡功能的异构体, 而3号外显子编码产物不含BH3功能域而不具有促凋亡功能[12, 13]。Ng等通过末端测序技术筛查慢性粒细胞性白血病患者的基因组时发现, BIM在2号内含子中发生了2 903个碱基对的缺失, 且该现象在非小细胞肺癌和慢性髓细胞白血病中与酪氨酸激酶抑制剂内源性耐药机制密切相关[5]。乳腺癌中HER2基因的失活可导致BIM基因的表达丰度增加, 同时BIM基因缺失影响Caspase凋亡因子的激活从而减少HER2失活引起的肿瘤退化; 这种HER2阳性乳腺癌亚型对特定BIM驱动突变的依赖性的现象被称为“ 致癌基因成瘾” [14] 。更多的临床数据证明在乳腺癌及多种肿瘤类型中, BIM缺失多态性为不良预后预测因子[4, 5, 6, 7, 8, 15, 16, 17, 18]。
本研究的336例乳腺癌患者中, 基因测序法检测出有39例患者为BIM基因缺失, 阳性率为11.61%; Snapshot 检测法发现47例患者为BIM基因缺失, 阳性率为13.99%。Ng等通过筛查2 597位健康个体发现BIM缺失多态性在亚洲人群中的发生率是12.3%, 但是在非洲及欧洲人群中却没有发现(0%)[5]; 人类基因组单倍体图(Hap Map)计划中的中国人BIM del携带率为20.5%, 这与本实验结果基本一致。Snapshot 法检测阳性率高于Sanger直接测序法。用配对卡方检验和Kappa检验分析两种检测方法的差异性和吻合度, 配对卡方检验结果为P=0.152, 两种方法的诊断结果差异无统计意义; 两种检测方法的吻合系数κ =0.695, P=0.000, 两种检测方法的结果有较好的总体一致性。但也存在部分患者只有Sanger直接测序法或Snapshot检测为阳性, 即两种方法之间在临床分析中可能存在一定的互补性。
两种方法所测BIM基因缺失存在一定的差异; Sanger测序法准确性较高但相对阳性率稍差, 考虑可能原因是长期石蜡包埋处理的组织DNA容易发生降解; Snapshot法阳性率相对Sanger测序法稍高, 其在降解肿瘤组织中更具有优势。两法均具有较高的灵敏度, Sanger测序法数据具有一定准确性和可靠性, 结合两种方法结果综合分析可以作为乳腺癌肿瘤BIM基因缺失多态性检测的有效方法, 为抗凋亡相关的临床试验研究或临床选用靶向治疗的决定提供参考数据。
本文初步回答了Sanger直接测序法与Snapshot法检测乳腺癌中BIM缺失吻合度情况; 我们进一步的工作在于回答BIM缺失突变与患者生存预后是否有关, 能否预测临床中特定治疗方案的疗效?针对上述BIM del多态性及凋亡信号通路异常引起的靶向治疗耐药能否通过BIM模拟物或BCL-2抑制剂, 包括ABT-737、Navitoclax/ABT-263和AT-101与乳腺癌靶向药物联合应用克服耐药?这些科学问题均有待于进一步研究。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|