作者简介:
蒋杰(1994-),女,四川广安人,硕士研究生,主要研究方向为肺癌免疫治疗。
目的 探讨SWI/SNF复合体(SWItch/Sucrose Non-Fermentable complex)不同亚基突变与晚期非小细胞肺癌(non-small cell lung cancer,NSCLC)的预后及与肿瘤细胞程序性死亡配体1(programmed death ligand 1,PD-L1)和肿瘤突变负荷(tumor mutation burden,TMB)表达的相关性。方法 回顾性收集2019年4月至2020年9月在广东省人民医院确诊为不可切除局部晚期或转移性NSCLC患者,采用二代测序的方法检测 ARID1A、ARID1B、SMARCA4、PBRM1、ARID2、SMARCB1、SMARCE1基因突变并计算样本TMB,免疫组化方法检测PD-L1的表达,结合患者的临床资料进行临床病理特征及生存分析。结果 在66例患者中,10例患者检测到同时存在2个亚基共突变,共突变率达15.2%;12例患者检测到SWI/SNF复合体亚基突变伴随 KRAS突变,共突变率达18.2%。 ARID1A、ARID1B、SMARCA4、PBRM1、ARID2、SMARCB1、SMARCE1亚基突变各占24%、12%、34%、12%、16%、1%、1%。SWI/SNF复合体亚基突变的NSCLC患者PD-L1≥1%占83.7%,其中 ARID1A和 SMARCA4突变患者PD-L1≥1%均占22.4%;各亚基突变的TMB具有统计学差异( P=0.01),其中 SMARCA4突变患者TMB≥10 Muts/Mb占24.2%。生存分析结果显示SWI/SNF复合体不同亚基突变之间的总生存时间差异无统计学意义( P=0.61)。结论 SWI/SNF复合体不同亚基突变在NSCLC中的预后无明显差异,但 SMARCA4突变倾向于伴随更高的TMB,其潜在的临床意义有待进一步研究。
Objective This study was designed to explore the prognosis of SWItch/Sucrose Non-Fermentable complex (SWI/SNF) complex subunits mutation and the relation with tumor programmed death ligand 1 (PD-L1) and tumor mutation burden (TMB) in unresectable locally advanced or metastatic non-small cell lung cancer.Methods Patients diagnosed with unresectable locally advanced or metastatic non-small cell lung cancer from April 2019 to September 2020 in the Guangdong Provincial People's Hospital were retrospectively collected. Next-generation sequencing (NGS) was used to detect ARID1A, ARID1B, SMARCA4, PBRM1, ARID2, SMARCB1, and SMARCE1 genomic alterations, TMB was calculated simultaneously, the expression of PD-L1 was detected through immunohistochemistry, and clinicopathological characteristics and survival analysis were carried out in combination with the clinical data of the patients.Results 15.2% (10/66) of patients were found to have two subunits mutation simultaneously; 12 patients (18.2%) were detected with SWI/SNF complex subunit mutations accompanied by KRAS mutation. ARID1A, ARID1B, SMARCA4, PBRM1, ARID2, SMARCB1, and SMARCE1subunit mutation accounted for 24%, 12%, 34%, 12%, 16%, 1%, and 1%, respectively. 83.7% of patients with SWI/SNF complex subunit mutation expressed PD-L1 ≥ 1%, and the expression of PD-L1≥1% accounted for 22.4% with ARID1Aand SMARCA4mutation, respectively; There was a significant difference in tumor mutation burden (TMB) between SWI/SNF complex subunits ( P=0.01), among which mutation of SMARCA4 accounted for 24.2% when TMB≥ 10 Muts/Mb. Survival analysis showed no significant difference in overall survival among different subunit mutations of the SWI/SNF complex ( P=0.61).Conclusions There was no significant prognostic of survival between different mutational SWI/SNF subunits in unresectable locally advanced or metastatic NSCLC. But patients with SMARCA4mutation tended to be higher TMB than others, which needs further investigation for its potential clinical value for immune therapy.
真核染色质包装状态、核小体组蛋白及DNA分子或空间结构发生改变称为染色质重塑。SWI/SNF(SWItch/Sucrose Non-Fermentable)复合体最早是在酿酒酵母(Saccharomyces cerevisiae)中发现的[1, 2], 是真核生物和原核生物中的一类进化保守的染色质重塑复合体和基因表达调控因子。它利用ATP水解产生的能量改变染色体的空间结构, 与DNA复制转录、DNA损伤修复、细胞分裂分化等过程有关[3]。SWI/SNF复合体有3种亚型:cBAF(canonical BRM/BRG1-associated factor), PBAF(polybromo-associated BAF), ncBAF(non-canonical BAF)[4]。cBAF和PBAF的核心亚基是SMARCC1、SMARCC2、SMARCD1/D2/D3和SMARCE1, SMARCA2(BRM)和SMARCA4(BRG1)是ATP酶, ARID1A/B和DPF1/2/3是cBAF的重要亚基, ARID2、PBRM1、PHF10和BRD7是PBAF的重要亚基[4, 5]。现有研究表明, SWI/SNF复合体突变与多种恶性肿瘤的发生发展相关, 包括非小细胞肺癌(non-small cell lung cancer, NSCLC)[4]、肾透明细胞癌(clear cell renal cell carcinoma, ccRCC)[6]、卵巢高钙血症型小细胞癌(small cell carcinoma of ovary-hypercalcemic type, SCCOHT)[7]、恶性横纹肌瘤(malignant rhabdoid tumor, MRT)[8]等, 但其对各种恶性肿瘤的肿瘤微环境和治疗疗效影响可能不同。
在全球, 肺癌发病率居于第二位, 死亡率居于第一位[9], NSCLC在肺癌中约占80%。目前针对表皮生长因子受体(epithelial growth factor receptor, EGFR)、间变性淋巴瘤激酶(anaplastic lymphoma kinase, ALK)等常见驱动基因靶点已经研发出许多有效药物并用于临床实践, 但对于其他非EFGR、ALK等驱动基因阳性患者的治疗仍然充满挑战。在本中心参与的CHOICE研究中发现, 131例肺腺癌患者中, TP53、EGFR、KRAS、STK11、SMARCA4突变率位于前五位, 而复合体亚基SMARCA4、ARID1A、ARID2突变率分别约为15%、6%、4%, 114例肺鳞癌患者ARID1A突变率约为7%, 上述现象在肿瘤基因组图谱(The Cancer Genome Atlas, TCGA)数据库中也得到了验证[10]。Zhou[11]的研究则显示PBRM1突变可能是NSCLC免疫治疗的负性预测生物标志物, Zhu[12]的研究显示ARID1A、ARID1B、ARID2亚基突变的NSCLC患者可能从免疫治疗中获益。并且在2021年世界卫生组织已将胸部SMARCA4缺失的未分化肿瘤列为一个新的肺肿瘤分类亚型[13]。由此可见SWI/SNF复合体在NSCLC中突变频率较高, 而不同亚基突变频率不尽相同, 并且不同亚基突变对NSCLC患者的预后影响可能不同, 因此本文针对SWI/SNF复合体不同亚基在NSCLC中的突变情况和预后意义以及免疫标志物程序性死亡配体1(programmed death ligand 1, PD-L1)表达水平和肿瘤突变负荷(tumor mutation burden, TMB)水平进行了回顾性分析研究。
本研究经医学伦理委员会批准(伦理号:GDREC2019400H), 回顾性收集2019年4月至2020年9月广东省人民医院诊断为不可切除局部晚期或转移性SWI/SNF复合体突变的NSCLC患者66例, 其中失访7例。所有患者的人口学资料包括性别、年龄、病理类型、吸烟状态、PD-L1表达水平和治疗方案等均从本院电子病历系统获取, 所有患者或家属均已知情同意并签署知情同意书。
总生存时间定义为从确诊为不可切除局部晚期或转移性NSCLC时间至死亡或随访截止时间。随访时间截至2021年9月30日。
使用吉因加公司的二代测序(next-generation sequencing, NGS)1 021 panel检测患者肿瘤组织或血浆的基因突变状态。
PD-L1蛋白表达水平的检测为免疫组织化学法, 采用Dako公司的22C3抗体或Roche公司的SP142抗体, 按肿瘤细胞阳性比例分数(tumor proportion score, TPS)判读PD-L1表达水平, 检测结果定义为阳性:≥ 1%, 阴性:< 1%。
TMB计算基于去除胚系突变后的体细胞突变数量, 单位为Muts/Mb, 计算规则是体细胞突变个数/编码区大小(1 021 panel编码区域为1 Mb)。高TMB定义为TMB ≥ 10 Muts/Mb, 低TMB定义为TMB < 10 Muts/Mb。
采用R软件(v4.1.2)进行数据分析, 计数资料采用频数和百分比表示, 组间比较采用Pearson卡方检验或Fisher精确检验分析SWI/SNF复合体不同亚基突变之间的PD-L1表达水平以及TMB的差异; 生存分析采用Kaplan-Meier法, Log-rank检验对P值进行检验, 当P< 0.05被认为差异具有统计学意义。
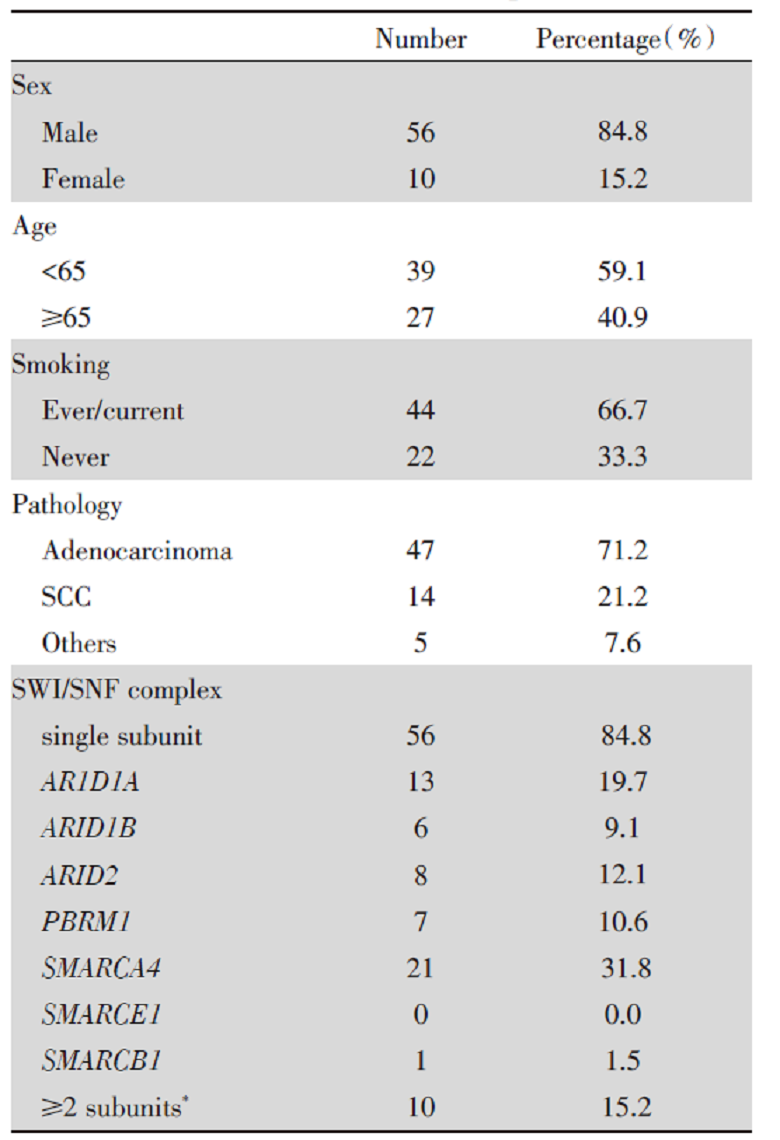
66例患者中, 男性占84.8%, 女性15.2%; 年龄31~86岁, 中位年龄62.5岁; 吸烟者44例, 从不吸烟者22例; 鳞癌14例, 腺癌47例, 其他5例包括腺鳞癌3例、大细胞神经内分泌癌2例, 见表1。
| 表1 SWI/SNF复合体突变的NSCLC患者的临床病理特征及亚基突变 Tab.1 The clinicopathological characteristics and subunits mutation of SWI/SNF complex in NSCLC |
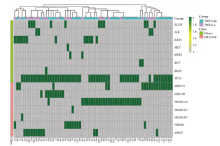
SWI/SNF复合体亚基突变包括ARID1A、ARID1B、ARID2、SMARCA4、PBRM1、SMARCE1、SMARCB1。其中, 11例SWI/SNF复合体突变患者伴随EGFR突变, 4例患者伴随ALK融合突变, 12例患者伴随KRAS基因突变, 2例患者伴随HER2 20外显子插入突变, 2例患者伴随RET融合突变, 1例患者伴随MET14外显子跳跃突变, 1例患者伴随BRAFV600E突变, 见图1。
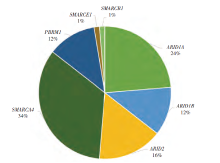
在66例NSCLC患者中, SMARCA4和ARID1A突变的患者较多, 分别有31.8%(21/66)和19.7%(13/66)的患者发生单个SMARCA4、ARID1A亚基突变, 10例患者检测到同时存在2个SWI/SNF复合体亚基突变, 即共突变率达15.2%(10/66), 见表1。而根据各亚基突变次数来看, 在SWI/SNF复合体单个亚基中, SMARCA4和ARID1A突变频率仍是较高的, 分别占比34%(26/76)、24%(18/76), ARID1B、ARID2、PBRM1、SMARCE1、SMARCB1分别占比12%(9/76)、16%(12/76)、12%(9/76)、1%(1/76)、1%(1/76), 见图2。
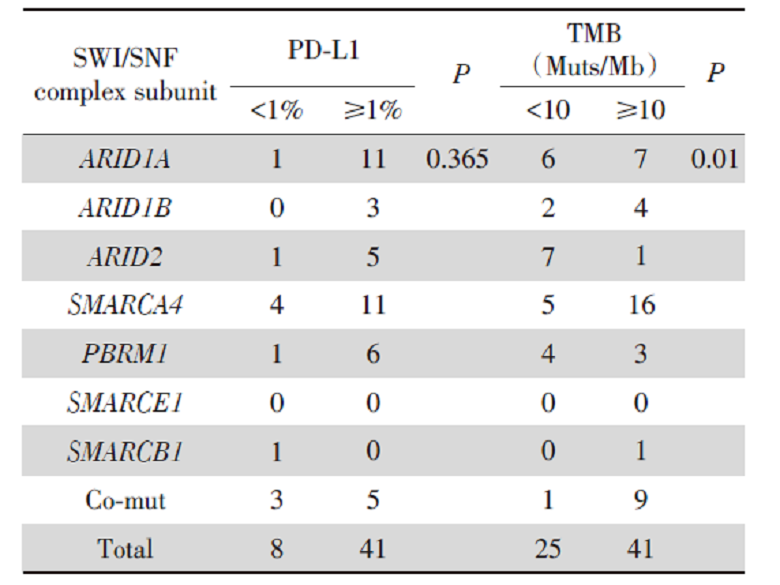
49例SWI/SNF复合体突变患者进行了PD-L1检测, 17例患者PD-L1未检测。SWI/SNF复合体突变的NSCLC患者PD-L1表达≥ 1%占83.7%, 其中ARID1A和SMARCA4突变患者PD-L1表达≥ 1%分别占22.4%和22.4%。SWI/SNF复合体各亚基突变患者之间的PD-L1表达差异无统计学意义(P=0.365), 见表2。
| 表2 SWI/SNF复合体亚基突变的PD-L1表达及TMB情况 Tab.2 The expression level of PD-L1 and TMB in SWI/SNF complex subunits |
66例SWI/SNF复合体突变患者进行了TMB的计算, TMB≥ 10 Muts/Mb占62.1%, TMB< 10 Muts/Mb占37.9%, 其中SMARCA4突变患者TMB≥ 10 Muts/Mb占24.2%。SWI/SNF复合体各亚基突变患者之间的TMB存在统计学差异(P=0.01), 且SMARCA4亚基突变倾向于伴随更高的肿瘤突变负荷, 见表2。
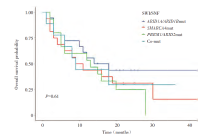
根据SWI/SNF复合体亚基的特征, 排除1例SMARCB1突变患者, 纳入58例患者并分为4组:ARID1A/ARID1B、SMARCA4、PBRM1/ARID2、Co-mut, 进行生存分析, 结果如图3所示, 各组的中位生存时间分别为13.6个月、9.4个月、12.8个月、9.4个月, SWI/SNF复合体不同亚基突变患者之间的总生存时间无统计学差异(P=0.61)。
SWI/SNF复合体属于ATP酶依赖的染色质重塑复合体家族(ATP-dependent chromatin remodeling complexes families, CRC families)成员之一[14], SWI/SNF复合体是重要的染色质重塑复合物, 由核心亚基、催化亚基和调节亚基组成, 通过重构核小体组蛋白的位置或组成成分, 可改变染色质的可及性及可访问性, 从而调控染色质的结构和基因表达[14]。现有研究主要是cBAF和PBAF复合体, ncBAF复合体是2018年才发现的复合体亚型[4]。在真核生物中, SWI/SNF复合体亚基不仅与启动子紧密结合, 还与其他调控区域紧密结合, 如增强子、DNA复制起始位点、DNA转录起始位点等, 同时发现SWI/SNF亚基与细胞核和细胞骨架中的某些蛋白也会结合, 从而影响分子相互作用[15]。
本研究是首次探索SWI/SNF复合体不同亚基突变之间与NSCLC的关系。在本研究NSCLC患者中, ARID1A和SMARCA4亚基突变发生率较高, 各占34%和24%, SWI/SNF复合体亚基共突变率为15.2%, 不同亚基突变之间的生存分析无统计学差异。Alessi[16]的研究显示, 在西方的NSCLC患者中, ARID1A和SMARCA4亚基突变率较高, 分别为28.1%、27.4%, SWI/SNF复合体亚基共突变率为14.1%, 与本研究的结果相似。另外, 相比SWI/SNF复合体野生型的NSCLC患者, SWI/SNF复合体亚基突变患者存在更低的驱动基因共突变率、总生存时间也更短[16]。而Jiang[17]通过分析TCGA和美国纪念斯隆凯特琳癌症中心(Memorial Sloan Kettering Cancer Center, MSKCC)数据库发现接受免疫治疗的ARID1A突变NSCLC患者比ARID1A野生型患者无进展生存期(progression-free survival, PFS)更长, 而PBRM1突变的NSCLC患者比PBRM1野生型患者的总生存时间(overall survival, OS)则更短。但两项研究均未探究SWI/SNF复合体不同亚基突变NSCLC患者之间的生存差异。
在不可切除局部晚期或转移性NSCLC中, 本研究结果显示SWI/SNF复合体不同亚基突变NSCLC患者之间的PD-L1表达无统计学差异, 但TMB有统计学差异(P < 0.05), 且ARID1A和SMARCA4突变患者PD-L1≥ 1%表达水平相对较高, SMARCA4突变患者的TMB相对较高。以往研究显示在NSCLC中, SWI/SNF复合体突变患者的PD-L1表达水平和TMB均较SWI/SNF复合体野生型患者高[16, 17, 18], 但并未研究SWI/SNF复合体不同亚基之间的PD-L1和TMB水平。
Alessi[16]的研究显示在接受免疫治疗的晚期NSCLC患者中, SWI/SNF复合体突变与野生型患者相比, PFS和OS没有明显的统计学差异, 但KRAS突变合并SMARCA4突变相比只有KRAS突变患者的PFS和OS更差。由此说明SWI/SNF复合体和KRAS突变可能共同影响肿瘤的免疫微环境, 但这还需要更多的研究去验证。SWI/SNF复合体亚基呈现高频突变, 尤其是在肿瘤和神经发育疾病中, 其突变率仅次于TP53[19]。因此, 在NSCLC中, 针对SWI/SNF复合体亚基与其他基因发生共突变进行深入的亚组分析可能为肿瘤精准治疗提供更多的依据。
综上所述, 在SWI/SNF复合体不同亚基突变的NSCLC中, SMARCA4和ARID1A亚基突变率较高, 各亚基突变PD-L1≥ 1%和TMB水平均较高, 但由于本研究的样本量较小, 因此未找到与预后明显相关的亚基突变。本研究仅探索了SWI/SNF复合体不同亚基突变NSCLC患者之间的生存差异, 而相比SWI/SNF复合体野生型NSCLC患者, SWI/SNF复合体突变是否会影响NSCLC患者的预后以及SWI/SNF复合体不同亚基突变如何影响NSCLC免疫表达谱仍待进一步研究。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|